E-learning 2. Analýza dat 2.2. Analýza vysokopokryvných genomických dat 2.2.1. DNA mikročipy 2.2.1.6. Normalizace
2.2.1.6.1. Proč normalizace?
Všechny laboratorní kroky mikročipového experimentu od návrhu po vytvoření souboru nezpracovaných dat jsou zdrojem technického vlivu na nezpracovaná data: druh vzorků (čerstvě zmrazený nebo fixovaný formalínem, tkáňové nebo krevní vzorky....), volba mikročipového sklíčka (cDNA, affymetrix...), DNA značení (typ fluorescenčních značek), hybridizace, skenování a software analýzy obrazu. Proto je nezbytné zkontrolovat, zda jsou nezpracovaná data ovlivněna nějakou náhodou nebo systematickými chybami. Normalizace je skupina transformací, jejímž cílem je tyto odchylky eliminovat. Jak tyto chyby nalézt a jak je opravit je popsáno v následujících částech. Při normalizaci bychom měli mít na paměti, že zde není jen nechtěná technická variabilita, kterou se pokoušíme detekovat a eliminovat, ale je zde také biologická variabilita, kterou se pokoušíme odhalit, a ta by eliminována být neměla. Cílem normalizace je vyloučit z dat nechtěné efekty a vytvořit data z různých mikročipových sklíček srovnatelné bez obav, že nalezené významnosti jsou kvůli technické odchylce.
Normalizace může být vykonána v rámci jednoho sklíčka (within array level) upravující technické odchylky uvnitř každého čipu, anebo mezi sklíčky (between array level) opravující distribuční rozdíly mezi různými čipy z našeho experimentu. Existují různé druhy technických odchylek, na každou z nich slouží speciální metody. Následující část popisuje metody rozpoznání typů přítomných odchylek v datech.
2.2.1.6.2. Diagnostické grafy
Vůbec první částí normalizace je identifikace odchylek. Nejlepší způsob je jejich vizualizace v diagnostických grafech. Zde vysvětlíme situaci pro dvoukanálové cDNA čipy, situace s jinými typy čipů jsou podobné a může být pro ně použita většina zmíněných grafů a metod.
Nejintuitivnějším způsobem, jak odhalit odchylky barviv, je bodový graf porovnávající oba samostatné kanály intenzit – červené barvivo a zelené barvivo. Jsou-li body rovnoměrně rozloženy kolem diagonály, pak zde žádné odchylky barviva nejsou:

Tento graf je ovšem schopen odhalit pouze lineární závislost. Avšak odchylky barviva jsou často nelineární. K odhalení nelineárního vztahu by měl být vykreslen MA graf. Je to bodový graf M-hodnot na ose x a A-hodnot na ose y. Kde M a A jsou definovány jako
![]() and
and ![]()
kde R a G reprezentují intenzity červeného a zeleného kanálu, v tomto pořadí. V mnoha experimentech je hlavním předpokladem, že většina genů neprojevuje v genové expresi mezi skupinami žádný rozdíl. Proto by většina bodů měla být rozložena na ose y kolem nuly, pokud není v intenzitách barviva žádný rozdíl:
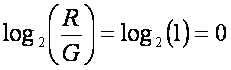
MA graf zřetelně odhaluje nelineární závislosti.

Obrázek: MA graf
Prostorové vzory mohou být vizualizovány pro obě intenzity - popředí a pozadí, ve formě hodnot log2 podílu pomocí vykreslení heat mapy. Jedná se o graf intenzit každého spotu uspořádaných podle jejich pozice na mikročipovém sklíčku:

Obrázek: Heat mapy hodnot log2 podílu: A) Čip bez jakékoli prostorové odchylky; B) Čip se systematickým jednostranným zkreslením
V horní části ukazuje heat mapa log2 podíly popředí a je vykreslena škálou barev od červené přes žlutou k bílé. Červená znamená, že jsou geny aktivní právě v jednom kanálu a bílá znamená, že jsou geny aktivní ve druhém. Žlutá barva znamená, že jsou intenzity stejné v obou kanálech. Každý gen je zobrazen barvou podle log2 podílu.
Pokud zde není žádná prostorová odchylka, měly by být barvy rozvrženy náhodně bez jakéhokoli viditelného vzoru. Tento graf může odhalit prostorové odchylky – prohlédněte si teplotní mapu nahoře vpravo.
Vykreslení heat mapy pro intenzity pozadí je hodnotné.
Dalším užitečným diagnostickým nástrojem je vykreslení krabicových grafů (boxplots). Skrze boxplot můžeme kontrolovat všechny intenzity buď mezi jednotlivými print-tips (jedná se o sázecí sady, které spotují sekvence na mikročipové sklíčko po čas jeho vytváření. Jsou uspořádány do bloků, proto se může vyskytnout rozdíl právě mezi bloky) v rámci každého čipu nebo intenzit mezi čipy. Po normalizaci by měla být průměrná intenzita a váha mezi print-tips srovnatelná, rovněž všechny intenzity mezi všemi čipy.
Každá normalizace by měla být kontrolována za pomocí vizualizace intensit před a po transformaci.
2.2.1.6.3. Normalizace v rámci jednoho mikročipového sklíčka
Normalizace v rámci jednoho sklíčka má za cíl vyrovnat technickou variabilitu uvnitř čipu. Základním předpokladem pro všechny normalizační metody je, že většina genů v rámci celého čipu nemá změny v expresi a že podíl intenzit je roven jedné. Tento předpoklad může být aplikován na čipy s velkým počtem genů. Pokud je jich pouze několik stovek, měli bychom být s normalizací opatrnější. Normalizace by měla být provedena v pořadí, jaké zde předkládáme.
cDNA čipy
Normalizace prostorových odchylek
Nejprve ze všeho bychom se měli zaměřit na úpravu prostorových odchylek. Často jsou způsobeny nestejnou hybridizací, tento problém může být na heat mapě zobrazen jako světlejší nebo tmavší oblasti ve středu nebo na okrajích. Jiná příčina prostorových výchylek může být print-tip problém. Sondy na mikročipových sklíčkách jsou obvykle sázeny (tištěny) skupinou malého počtu jehlel a každá z nich vytváří na čipu obdélník spotů zvaný print-tip. Jestliže je nějakým způsobem některá z těchto jehel zničena, odpovídající print-tip spoty se mohou od zbývajících lišit v hybridizaci. Na druhé straně jsou situace, ve kterých heat mapa ukazuje jasné prostorové vzory, které ovšem není vhodné normalizovat. Dokonce i když je doporučeno rozdělit geny na čipu náhodně, jestliže z nějakého důvodu má mikročipové sklíčko geny uspořádané podle jejich biologické funkce, může to na heat mapě vytvářet očividné vzory. Takové vzory by neměly být normalizovány. Často jsou obvyklým způsobem na čip naneseny kontrolní spoty, aby kontrolovaly jakoukoli prostorovou odchylku. Tyto spoty mohou obsahovat spike kontroly, provozní geny nebo vůbec žádné sondy.
Další důvod prostorových odchylek může být nestejnoměrné omytí čipu nebo špatné vložení čipu do skeneru, což způsobuje rozdílné intenzity fluorescence napříč čipem.
Jednou z nejlepších metod sloužících k eliminaci prostorových odchylek je lowess nebo také loess regrese (Locally Weighted regression). Je-li lineární funkce použita k odhadnutí trendu s lokální regresí, pak se to nazývá lowess. Aplikuje-li se kvadratická funkce, mluvíme o loess regresi. Loess funkce je na data použita stupňovitým způsobem k odhadnutí lokálních trendů, pomocí metody posuvného okna. To má za následek vyhlazení křivky, která by měla odhalovat skrytý trend. Odhadovaná křivka je pak odečtena od původních hodnot.
Normalizace pozadí
Normalizace pozadí by měla následovat po prostorové normalizaci. V oblasti mezi spoty je vždy nějaká redundantní fluorescence, kterou je velmi důležité odstranit od skutečných intenzit spotů popředí.
Jestliže předpokládáme, že tento efekt pozadí je aditivní, pak intenzity pozorovaného spotu jsou součty intenzity pozadí a skutečného signálu, takže nejjednodušší přístup k odhadnutí skutečného signálu je odečíst signál pozadí (šumu) od pozorovaného signálu. Je zde ovšem riziko získání negativních intenzit. Např. když mřížka během obrazové analýzy nesedí s maximální přesností na všechny spoty, pak signál pozadí může být zkreslen pixely, které obyčejně náleží spotu popředí. V takových případech mohou být intenzity pozadí dokonce vyšší než popředí a není zde žádné logické vysvětlení negativních intenzit. Tento problém může být ustálen odečtením průměru všech prázdných spotů na čipu, namísto lokálních pozadí.
Nicméně někteří autoři mají za to, že efekt pozadí není aditivní, a někteří se domnívají, že pozadí by nemělo být zcela odstraněno ze spotového signálu popředí.
Normalizace odchylek barviva
Třetím krokem normalizace v rámci jednoho sklíčka by měla být úprava efektu barviva. Současné cDNA mikročipové technologie používají k rozlišení vzorků dvě různá barviva. Ačkoli mají tato dvě barviva mnoho společného, jsou zde nepatrné rozdíly, které mohou znemožnit přímé porovnání. Prvním rozdílem je různá afinita molekul barviva na DNA a druhým je odlišnost v excitaci použitím UV skeneru. Rozdíly v označení barviva jsou nejčastějším zdrojem odchylek, ale na druhé straně je lze také snadno identifikovat. Jeden způsob, jak se vypořádat s tímto problémem, je design vyměněných barviv. To ovšem vyžaduje dvakrát větší množství čipů, což je docela nákladné, hlavně když existují statistické metody, které mohou tyto rozdíly vyrovnat.
Vyhlazení dat je prováděno na datech dříve uvedených v MA bodovém grafu. V první řadě je odhadována přímka shodných expresí. Otázkou je, která data bychom měli pro tento odhad použít. Obvykle se používá invariantní sada kontrolních spotů jako jsou provozní geny (referenční geny), u nichž se očekává, že mají podobnou expresi v celé skupině. Problém je, že kontrolní spoty nejsou přítomné na každém čipu nebo je jejich počet pro regresi příliš malý. Podle předpokladu, že většina genů má konstantní expresi napříč všemi klinickými skupinami, které právě porovnáváme, můžeme ze všech genů na čipu tuto křivku odhadnout. Pro odhad křivky existuje několik metod, například můžeme zase použít loess regresi nebo metody založené na křivkách.
Podobně jako v prostorové normalizaci je pak odhadovaná křivka odečítána od původních hodnot a efekt normalizace je kontrolován na před/po diagnostických grafech:

Obrázek: MA grafy před a po normalizaci barviv
Jak jsme již zmínili na začátku této kapitoly, kroky normalizace by měly být aplikovány v uvedeném pořadí. Kdybychom použili normalizaci barviv před prostorovou normalizací, mohly by prostorové efekty vážně zkreslit efekty barviv, a to by mohlo vést k nesrovnalostem.
2.2.1.6.4. Normalizace mezi sklíčky
Ačkoli jsou všechny čipy skenovány stejným skenerem a máme v rámci každého čipu upraveny technické vlastnosti, není distribuce intenzit signálu mezi čipy stejná. Je to důsledkem biologických rozdílů mezi vzorky, ale především důsledkem různých množství mRNA a mírně odlišné manipulace se vzorky, dokonce i když je proces v laboratoři dobře standardizován. Srovnatelnosti intenzit mezi čipy může být dosaženo mezi normalizovanými čipy. Během této normalizace se pokoušíme vyrovnat distribuci intenzit všech čipů tím, že vycentrujeme mediány a nastavíme váhy na stejnou hodnotu.
Tento druh normalizace může vysvětlovat skupiny (podmínky), které právě porovnáváme, většinou když očekáváme mezi porovnávanými skupinami významné rozdíly. U normalizace v rámci podmínek normalizujeme čipy s replikovanými vzorky podle stejné podmínky (ve stejné skupině) a během normalizace napříč podmínkami, čipy jsou normalizovány mezi skupinami. Při aplikaci této poslední normalizace musíme být opatrní, abychom neeliminovali biologickou variabilitu.
Jedna z nejlepších metod sloužící k tomuto účelu je kvantilová normalizace. Tato metoda může normalizovat oba typy kopií a srovnat je na stejnou váhu. Je to založeno na pořadí pozorování, takže neexistují žádné předpoklady na rozložení dat. Nejprve je normalizace založená na pořadí provedena na podskupině invariantních genů, a pak jsou lineární interpolací odhadovány zbývající intenzity spotů. Medián a váhy intenzit normalizovaných čipů mohou být porovnány pomocí vykreslení box-plotů:

Obrázek: Čtyři čipy před a po kvantilové normalizaci