E-learning 2. Analýza dat 2.2. Analýza vysokopokryvných genomických dat 2.2.1. DNA mikročipy 2.2.1.11. Analýza biologických drah
2.2.1.11.4. Konkrétní metody
V předešlé části jsme probírali mnoho obecných problémů, s nimiž se při provádění analýzy genových sad setkáme. Mnoho z nich po nás požaduje, abychom provedli volbu (Použijeme kompetitivní metodu? Použijeme metodu závislou na dělící hranici?) a kombinace těchto voleb nás vede k širokému okruhu potenciálních analytických nástrojů. V nedávném článku Huang et al. [2009] je vyjmenováno 68 takových metod a to ještě tento seznam není úplný. Pokrýt všechny metody v rozsahu tohoto úvodního textu nelze, zaměříme se tedy na malý výběr nástrojů, což umožní čtenáři okusit různé druhy softwarů analýzy genových sad.
2.2.1.11.4.1 TopGO
Stejně jako mnoho nástrojů pro analýzu mikročipů je TopGO [Alexa et al., 2006] dostupný v R-knihovně z Bioconductor projektu. Jak již název signalizuje, program analyzuje GO-termíny. TopGo nabízí dva základní statistické testy, které jsme již dříve rozebírali: Fisherův exaktní test a kompetitivní Kolmogorův-Smirnovův test. Vylepšenou vlastností programu je, že bere do výpočtu hierarchickou strukturu GO databáze. Když je termín potomka (např. "accute inflammatory response") vysoce významný, bude to automaticky dělat významné také jeho rodičovské uzly (např. ”inflammatory response”), což způsobuje redundantní výsledky. TopGO nabízí ”bottom-up” (zdola nahoru) analytický přístup, kde jsou geny významných termínů potomka v analýze jejich rodičů eliminovány, např. ”inflammatory response” by bylo významné pouze pokud by také geny, které již nejsou spojeny s ”accute“ termínem potomka, byly dostatečně odlišně exprimovány. Navíc TopGO ke svým analytickým vlastnostem také umožňuje grafické znázornění nejvýznamnějších GO-termínů do podoby barevně značeného stromu.
2.2.1.11.4.2 GSEA (Gene Set Enrichment Analysis)
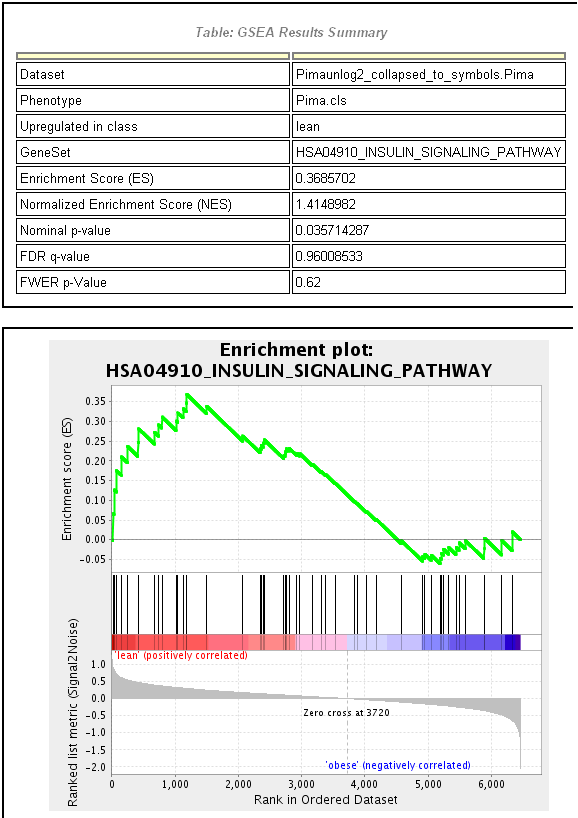
GSEA navržena Subramanian et al. [2005] se stala v posledních letech velmi populárním nástrojem. Vyvinul ji Broad Institute spolu s MSig databází. Je dostupná jako samostatný analytický program, ale také může být zpřístupněna jinými způsoby (např. z prostředí R). Jádro původního GSEA algoritmu je nepatrně pozměněnou verzí kompetitivního Kolmogorova-Smirnovova testu. Odpovídající testová statistika se v GSEA terminologii nazývá „skóre obohacení“. GSEA není založena jen na p-hodnotách, ale také umožňuje k seřazení genů použít jiné hodnoty sumarizující data (t-statistiku, hodnoty fold change, SNR). K výpočtu p-hodnoty nabízí jak převzorkování čipů, tak genů. Na obrázku 2.2.1.11.6 vidíme výstup GSEA pro nejvýznamnější genovou sadu třídy C2 (vytvořené genové sady z online databází drah) v MSigDB. Jedná se o insulinovou signalizační dráhu, velmi věrohodný výsledek, protože obézní Pima Indiáni mají sklon trpět diabetem typu 2, nemocí, která je způsobena poruchou této dráhy. Graf na výstupu je podobný druhu grafu na obrázku 2.2.1.11.5 (b), ale otočený o 45 stupňů.
Obrázek 2.2.1.11.6: GSEA výstup pro lidskou signalizační dráhu insulinu
2.2.1.11.4.3 GenMAPP a PathVisio
Zatímco se programy jako TopGO a GSEA zaměřují na analýzu genových sad a nabízí k tomuto účelu své vyvinuté algoritmy, GenMapp [Salomonis et al.,2007] a PathVisio [van Iersel et al., 2008] jsou hlavně vizualizačními nástroji drah. Oba projekty pracují společně na sbírání map drah, které pak mohou být spojovány s mikročipovými daty. PathVisio je úzce spjato s WikiPathways projektem a podobně jako GenMapp umožňuje barevné ztvárnění mapy dráhy podle mikročipových dat, jak je vidět na obrázku 2.2.1.11.3. Lze například vybrat, že všechny významné geny s hodnotou foldchange vyšší než 1,5, budou zvýrazněny červeně, což poskytuje okamžitý přehled, které komponenty drah jsou odlišně exprimovány. Oba programy také zahrnují nějaké analytické možnosti, hlavně modifikace Fisherova testu.
2.2.1.11.4.4 Více statisticky zaměřené nástroje
Poslední dva příklady, které bychom rádi představili, jsou sofistikovanější statistické metody. Global Test poprvé uvedl Goeman et al. [2004]. Oproti většině metod, které jsme popisovali, nepoužívá na vstupu sumarizaci dat jako je p-hodnota, ale používá všechny expresní hodnoty genů v genové sadě. Další neobvyklá vlastnost je, že zaměňuje obvyklou roli vysvětlujících proměnných a odpovědí. Všechny jiné metody, které jsme dosud probírali, se pokouší vysvětlit genovou expresi pomocí dvou skupin (např. hubení a obézní). Global Test se pokouší vysvětlit skupiny pomocí genové exprese, tj. v této dvou-skupinové situaci je to podobné jako s modelem logistické regrese s tím rozdílem, že test nedává p-hodnoty pro jednotlivé geny, ale udává skóre celé genové sady. Na rozdíl od zmiňovaných metod, se tato metoda může také zabývat jinými komplexnějšími návrhy, než je jednoduché porovnávání dvou tříd, např. lze ji použít v klinických studiích na asociované genové sady s dobou přežití. Srovnatelný flexibilní nástroj je Global ANCOVA, kterou poprvé představili Mansmann a Meister [2005]. Tento program opět používá původní hodnoty exprese a nepoužívá sumarizace dat. Hlavním rozdílem od Global Test je, že používá klasické nastavení, co se týká genové exprese jako odezvy, která je vysvětlována návrhem experimentu. Oba tyto nástroje jsou dostupné z prostředí Bioconductor v knihovnách globaltest a GlobalAncova.