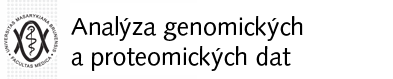E-learning 2. Analýza dat 2.2. Analýza vysokopokryvných genomických dat 2.2.1. DNA mikročipy 2.2.1.11. Analýza biologických drah
2.2.1.11.2. Zdroje biologických drah a genových sad
Sdílení informací na internetu je základním stavebním kámenem bioinformatiky. Sekvenční a proteinové databáze jsou nejstarší a nejznámější z těchto zdrojů a poskytují detailní informace o jednotlivých genech a proteinech. Databáze, které budeme rozebírat v této části, nahlíží na věc opačným směrem: ukládají geny v obecnějším kontextu, nabízejí skupiny a kategorie genů, které mají něco společného.
2.2.1.11.2.1 Gene ontology databáze
Srdcem Gene Ontology (GO) project [Consortium, 2004] je řízený slovník (ontologie), který je tvořen tzv. GO-termíny. Tyto termíny jsou děleny do tří hlavních kategorií: buněčná komponenta, biologický proces a molekulární funkce. Tyto tři termíny tvoří vrchní uzly hierarchické databáze, která je dostupná na http://amigo.geneontology.org/cgi-bin/amigo/go.cgi. Každý GO-termín v databázi může mít rodičovské uzly (obecnější termíny) a uzly potomka (specifičtější termíny). Pokud v GO databázi vyhledáváme termín „inflammation“ (zánět), pak zjistíme, že správný termín je „inflammatory response” (zánětlivá odpověď) a má identifikátor ”GO: 0006954” (Obrázek 2.2.1.11.1). Při pohledu na hierarchii vidíme, že jako nejvyšší rodičovský uzel je „biological process“ (biologický proces) a dva přímí rodičové jsou „response to wounding” (odpověď na zranění) a „defense response” (obranná odpověď), zatímco přímý uzel potomka je pouze „acute inflammatory response” (akutní zánětlivá odpověď).
Obrázek 2.2.1.11.1 Screenshot: GO databáze vyhledávající „inflammation“
Vidíme, že tomuto termínu odpovídá 1173 genových produktů, což se zdá být velké množství. Ale měli bychom mít na paměti, že GO není specificky navržena pouze pro jeden organismus, ale pokouší se zachytit biologické znalosti na velmi obecné úrovni. Geny, které jsou spojeny se „zánětem“ („inflammation“) myši, krysy, člověka atd. tedy budou počítány několikrát. Také stojí za povšimnutí, že genové produkty mohou být jak geny, tak proteiny. Při použití například Bioconductor balíku annotate je možné rozpoznat všechny sady sond na Affymetrix čipu, které odpovídají jistému GO-termínu. V našem příkladě HGu95e-Array a termínu inflammation (zánět) jsme našli na čipu 96 genů, tj. pouze malý zlomek z 1173 genových produktů. Stojí za zmínku, že jak jiné online zdroje, tak je i GO databáze stále pozměňována. Než se vytvořil první návrh tohoto článku, se zde udávaná čísla již změnila a budou zase jiná, jakmile čtenář tohoto článku zkusí naše vyhledávání provést znovu.
2.2.1.11.2.2 KEGG databáze biologických drah
I když je GO databáze velmi informativní a široce užívaná v mikročipové analýze, znamená její univerzální přístup, že nemůže zachytit specifičtější biologické znalosti. Biologické dráhy jsou koncept, který se pokouší popsat biologické mechanismy mnohem detailnějším způsobem. Dráha neposkytuje pouze seznam genů odpovídajících biologickému procesu, ale také podrobně uvádí grafickými způsoby vztahy mezi geny v buňce. Tento typ dráhy se často nazývá signalizační dráha (signaling pathway) nebo genová regulační síť (gene regulatory network). Slovo dráha se na druhou stranu také používá k popisu interakcí mezi metabolity (metabolické dráhy) nebo proteiny (proteinové dráhy), což někdy vede ke zmatení.
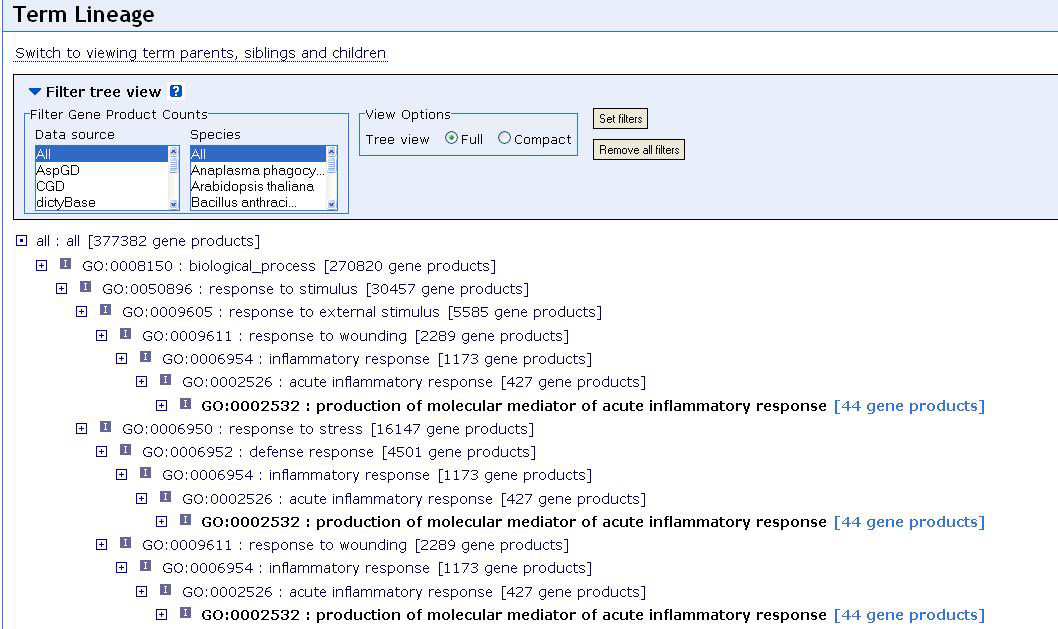
KEGG (The Kyoto Encyclopedia of Genes and Genomes) je projekt, který mimo jiné zajišťuje přístup k databázi drah na http://www.genome.jp/kegg/pathway.html [Ogata et al., 1999]. Oproti GO jsou informace získané z KEGG detailnější a k organismům specifičtější. Na obrázku 2 je vidět příklad mapy lidské signalizační dráhy insulinu. Nezobrazuje pouze zapojené geny, ale díky šipkám znázorňuje stupňovitou regulaci v buňce. Stojí za povšimnutí, že symboly zobrazené na mapě nejsou vždy geny, ale často také proteiny, které kódují nebo celé skupiny genů. Například vidíme, že gen INSR (receptor insulinu) reguluje obdélník zvaný SHC. Pokud klikneme na tento obdélník, zjistíme, že ve skutečnosti tento obdélník představuje skupinu příbuzných genů zvaných SHC1-SHC4.
Opět jsou zde Bioconductor nástroje (například knihovna KEGG.db), které umožňují přístup do této databáze z prostředí R. Většina analytických nástrojů přesto žádné struktury dráhy nepoužívá, využívá pouze seznam genů obsažených v dráze. U těchto druhů analýzy, když přijde na statistickou analýzu, se dráha od GO-termínu moc neliší: obojí definuje skupinu genů.
Obrázek 2.2.1.11.2: Signalizační dráha insulinu znázorněna KEGG databází
2.2.1.11.2.3 Wikipathways
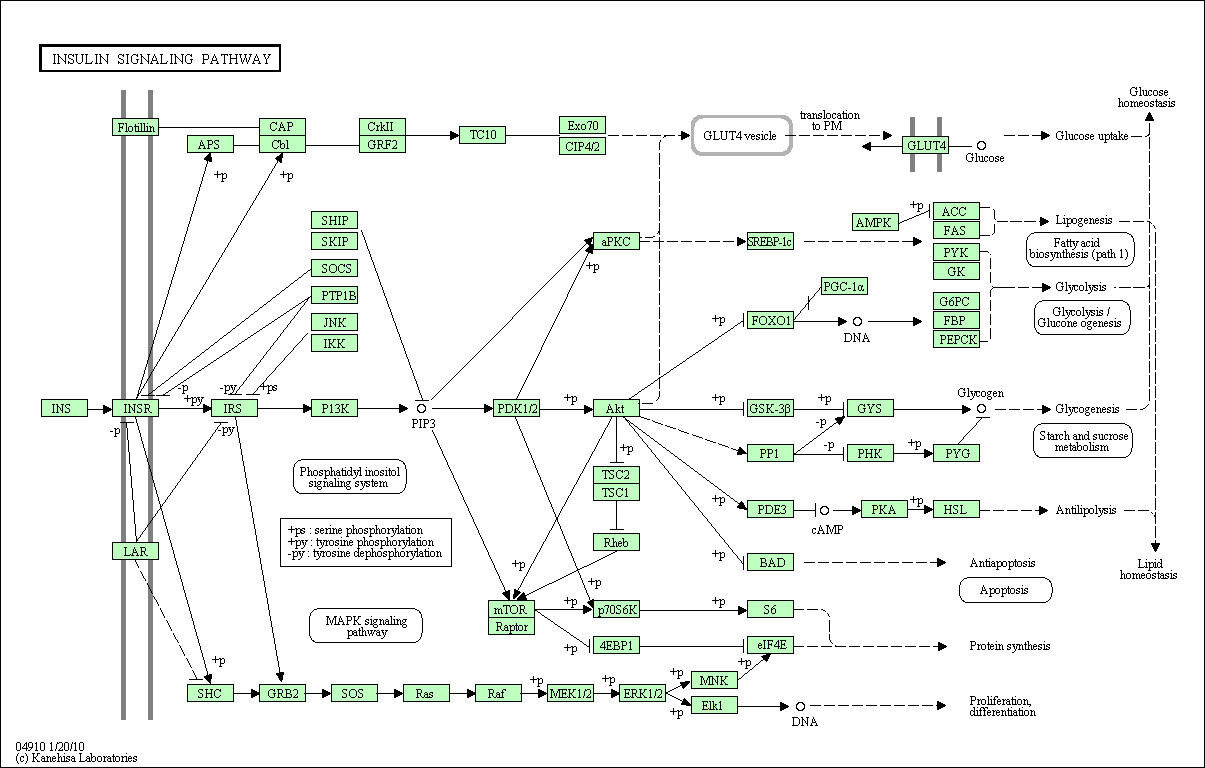
Tak jak všechny databáze má i KEGG problém držet krok se stále se vyvíjejícími biologickými znalostmi v literatuře a jiných databázích. Wikipathways project [Pico et al., 2008] se pokouší s tímto problémem vypořádat tím, že používá podobný dynamický přístup ke svému obsahu jako Wikipedie. Jednoduchým vstoupením na webovou stánku http://www.wikipathways.org může kterýkoli biolog uvnitř této databáze přidávat nové dráhy nebo upravovat již existující. Cílem je zaměřit se na definice drah, které jsou obecně přijímány vědeckou komunitou. Na obrázku 2.2.1.11.3 lze vidět signalizační dráhu insulinu v podání WikiPathway. Jak můžeme vidět, znázornění je jednodušší, než u mapy KEGG dráhy, což na druhou stranu umožňuje, že je tento formát jednodušší k editaci. S Wikipathways nejsou spojeny žádné Bioconductor nástroje, ale všechny existující dráhy lze stáhnout v různých formátech.
F)Obrázek 2.2.1.11.3: Signalizační dráha insulinu v podání WikiPathway
2.2.1.11.2.4 MSigDB (The Molecular Signature Database)
Jak bylo řečeno, GO databáze a databáze drah jako KEGG nebo Wikipathways jsou založeny na koncepčně velmi odlišných formulacích biologických znalostí, ale z hlediska analýzy vedou ke stejné otázce: jak můžeme rozhodnout, zda seznam genů odpovídajících dráze nebo GO-termínu je odlišně exprimovaný? MSigDB [Subramanian et al., 2007] to bere v potaz a používá obecnější pojetí genových sad. Tyto genové sady jsou děleny do pěti hlavních tříd (C1-C5):
- C1: Poziční genové sady pro každý lidský chromozom a cytogenetickou skupinu.
- C2: Vytvořené genové sady z online databází biologických drah, publikací v PubMed a znalostí expertů v této oblasti.
- C3: Tematické genové sady založené na konzervativních cis-regulačních tématech ze srovnávací analýzy.
- C4: Vypočítané genové sady určené expresí sousedících genů, zaměřeno na 380 genů spojených s rakovinou.
- C5: GO genové sady skládající se z genů anotovaných stejným GO-termínem.
Vidíme, že ačkoli se naše dříve zvažované typy genových sad objevují v C2 a C5, má MSigDB mnohem širší pohled na to, co může genovou sadu zájmu definovat.